Methylcitratzyklus
Vorhaben und Ziele
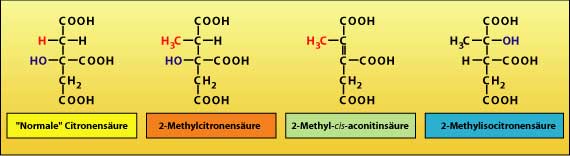
Der Methylcitratzyklus (MCC) wurde im Jahr 1974 erstmals durch die Arbeitsgruppe von Tabuchi als ein Stoffwechselweg der Propionatverwertung in Hefen beschrieben. In den letzten Jahren zeigte sich, besonders durch die Zugänglichkeit umfangreicher Sequenzdaten, daß die Gene des MCC bei Gram-negativen Bakterien weit verbreitet sind. Im Rahmen unserer Untersuchungen zum Abbau von Lävulinsäure in dem chemolithoautotrophen Bakterium Ralstonia eutropha konnte nachgewiesen werden, daß dieses Bakterium die kurzkettige Fettsäure Propionsäure ebenfalls über den MCC verstoffwechselt. Die Gene des MCC wurden kloniert und die Nukleotidsequenzen bestimmt; es zeigte sich, daß sie ein Gencluster bilden, welches aus den Genen prpR, prpB, prpC, acnM, ORF5 und prpD besteht. Das Translationsprodukt von prpC katalysiert den einleitenden Schritt des MCC, die Verknüpfung von Propionyl-CoA mit Oxalessigsäure zur 2-Methylcitronensäure. Diese wird zur 2-Methyl-cis-aconitsäure dehydratisiert und dann zur 2-Methylisocitronensäure hydratisiert. An dieser Isomerisierung sind die Genprodukte von acnM und ORF5 beteiligt. Diese ersten Schritte des MCC verlaufen analog zu den einleitenden Schritte des Citronensäurezyklusses. 2-Methyl-isocitronensäure wird dann durch eine Methylisocitronensäure-Lyase (prpB-Genprodukt) in Succinat und Pyruvat gespalten; diese Substanzen werden anschließend durch den "normalen" Stoffwechsel genutzt. Das prpR-Genprodukt ist an der Regulation des MCC beteiligt. Der MCC wird außer in R. eutropha HF39 auch noch in Burkholderia sacchari IPT101T, Pseudomonas putida KT2440 und anderen Mikroorganismen untersucht. |
Abb. 1 Der Methylcitratzyklus |
 |
Weiterhin wurde ein zweites prpC-Gen (prpC2) identifiziert, welches in der Nähe mit Genen liegt, die für Enzyme des Citronensäurezyklus codieren (Malat-Dehydrogenase, Citrat-Synthase, Succinat-Dehydrogenase, Aconitase). Zugleich wurden weitere Gene kloniert und charakterisiert, die am Abbau von Lävulinsäure beteiligt sind. Unter anderem ein potentielles Lävulinat-Reduktase-Gen levR. Ziel sollte hier sein, durch gezielte Veränderung des Stoffwechselflusses biologisch abbaubare Kunststoffe herzustellen, die als Hauptbestandteil 4-Hydroxyvaleriansäure enthalten. Der Citronensäurezyklus und der damit assoziierte Glyoxylatzyklus stellen zentrale Drehscheiben des Stoffwechsels dar und sind somit wichtige Ziele für die Konstruktion genetisch-modifizierter Stämme von R. eutropha, die zur fermentativen Produktion von z. B. Polyhydroxyalkanoaten oder 2-Methylcitronensäure und 2-Methylisocitronensäure genutzt werden können. Ein weiterer Aspekt dieses Projektes liegt daher in der Klonierung und Expression der Gene des Citronensäure- und Glyoxylatzyklus sowie der Erzeugung von Interposon-Mutanten und der phänotypischen Charakterisierung dieser Mutanten.
Methoden und Strategien
Die Untersuchungen des Methylcitronensäurezyklus erfolgen mittels molekulargenetischer Methoden (PCR, RT-PCR, Transposonmutagenese, Interposon-Mutagenese, DNA-Sequenzierung, Southern-Hybridisierung, Koloniehybridisierung, etc.), proteinchemischer Methoden (heterologe Expression, His-Tag-Fusionsproteine, Enzymassay, Proteinmodelling, 2-D-Gelelektrophorese etc.) sowie spektroskopischer und chromatographischer Methoden (HPLC, GC, GC/MS).Weiterhin können durch die Kenntnisse des bakteriellen Metabolismus und die Zugänglichkeit von Daten aus dem Ralstonia eutropha Genomprojekt sowie anderen Genomsequenzierungsprojekten neue Stoffwechselwege etabliert oder bestehende modifiziert werden (metabolic engineering), um die Produktion von 2-Methylcitronensäure und 2-Methylisocitronensäure zu steigern.
Stichwörter:
Propionsäure-Metabolismus, Citronen-/Methylcitronensäurecyclus Methylcitronensäure-Synthase, Methylisocitronensäure-Lyase, Methylcitronensäure, Methylisocitronensäure, Ralstonia eutropha, Burkholderia sacchari, Pseudomonas putida| Am Projekt beteiligte Mitarbeiter | Koorperationspartner, Förderung, Finanzierung | |
| Zur Zeit: Früher: | Christian Ewering Alexander Steinbüchel Axel Bethke Nadine Bruland Christian Brämer Florian Heuser Zhengxiang Wang | Westfälische Wilhelms-Universität Münster |
Publikationen
I Publications in peer reviewed journals (original contributions)
- Ewering, C., C. O. Brämer, N. Bruland, A. Bethke, and A. Steinbüchel. 2005. Three tricarboxylate synthases in Ralstonia eutropha: one citrate synthase and two 2-methylcitrate synthases. Appl. Microbiol. Biotechnol. DOI 10.1007/s00253-005-0009-2.
- Wang, Z.-X., C. O. Brämer, and A. Steinbüchel. 2003. The glyoxylate bypass of Ralstonia eutropha. FEMS Microbiol. Lett. 228:63-71.
- Brämer, C. O., and A. Steinbüchel. 2002. The malate dehydrogenase of Ralstonia eutropha and functionality of the C3/C4-metabolism in a Tn5-induced mdh mutant. FEMS Microbiol. Lett. 212:159-164.
- Brämer, C. O., L. F. Silva, J. G. C. Gomez, H. Priefert, and A. Steinbüchel 2002. Identification of the 2-methylcitrate pathway involved in the catabolism of propionate in the polyhydroxyalkanoate producing bacterium Burkholderia sp. IPT-101 and analysis of a mutant accumulating a copolyester with higher 3-hydroxyvalerate content. Appl. Environ. Microbiol 68:271-279.
- Brämer, C. O., and A. Steinbüchel. 2001. The methylcitric acid pathway in Ralstonia eutropha: new genes identified in propionate metabolism. Microbiology 147:2203-2214.
- Brämer, C. O., P. Vandamme, L. F. da Silva, J. G. C. Gomez and A. Steinbüchel. 2001. Burkholderia sacchari, sp. nov., a polyhydroxyalkanoate-acumulating bacterium isolated from soil of a sugar-cane plantation in Brazil. Int. J. Syst. Evol. Microbiol. 51:1709-1713.

