Erste 3D-Struktur von Regulator-Protein aufgedeckt
Proteine sind unverzichtbare Bestandteile lebender Organismen. Sie sind nicht nur eine „Bausubstanz“ für den Körper, sondern ermöglichen beispielsweise die molekulare Kommunikation der Zellen, sind für das Entstehen von Nervenimpulsen nötig und kontrollieren chemische Reaktionen. Entscheidend für die Funktion der Proteine ist ihre dreidimensionale Struktur. Ist diese bekannt, können Rückschlüsse auf die Funktionsweise von Proteinen gezogen werden. Ein Team um Prof. Dr. Daniel Kümmel von der Westfälischen Wilhelms-Universität (WWU) Münster und Prof. Dr. Stefan Raunser vom Max-Planck-Institut (MPI) für molekulare Physiologie in Dortmund hat nun die Struktur eines Proteinkomplexes aufgeklärt, der ein wichtiger Regulator von zellulären Abbauprozessen ist.
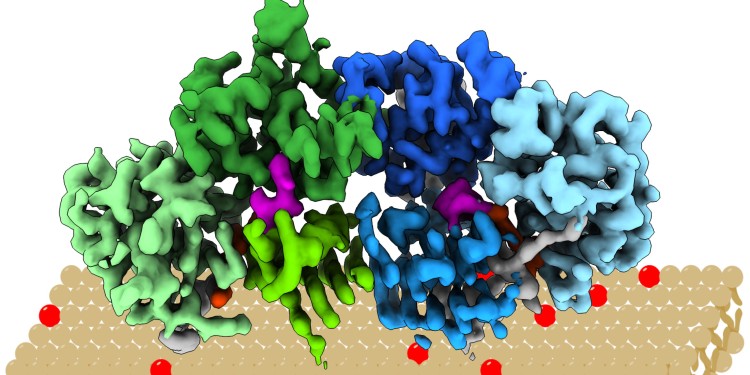
Dieser Proteinkomplex mit der Bezeichnung „Mon1/Ccz1“ bestimmt, welche intrazellulären Vesikel ihren Inhalt an den zellulären „Recyclinghof“, das Lysosom, liefern. Dazu dockt er an die Vesikelmembran an und ergänzt dort eine Markierung. Intrazelluläre Vesikel sind Membranbläschen, die Material durch die Zelle transportieren. Im Lysosom wird ihr Inhalt abgebaut und wiederverwertet. Durch die Aufklärung der Struktur in nahezu atomarer Auflösung konnten die Forscher nun unter anderem klären, wie der Proteinkomplex die passenden Vesikel erkennt. So zeigten sie zum Beispiel, dass der Komplex einen positiv geladenen, relativ flachen Bereich hat, der seine Orientierung nach dem Andocken an die Vesikelmembran bestimmt.
„Mon1/Ccz1 gehört zu einer Familie von Regulatoren, für die bisher keine strukturellen Informationen vorliegen. Diese Komplexe sind in vielfältige zelluläre Vorgänge eingebunden und teilweise auch mit dem Auftreten von Entwicklungsstörungen wie Albinismus und Blutgerinnungsdefekten assoziiert“, unterstreicht Daniel Kümmel. „Unsere Struktur liefert nun eine Grundlage, um diese Zusammenhänge besser zu verstehen.“
Der untersuchte Proteinkomplex stammt aus dem Fadenpilz Chaetomium thermophilum und ist unter Laborbedingungen besonders leicht zu handhaben und stabil. Er kann als Modell für die humanen Proteine dienen. Zur Bestimmung der Proteinstruktur setzten die Wissenschaftler hochauflösende Kryoelektronenmikroskopie ein. „Mit dieser Methode können wir die Struktur von Proteinen bei Temperaturen um minus 150 Grad Celsius im nahezu natürlichen Zustand untersuchen“, sagt Stefan Raunser. Die Forscher überprüften ihre Ergebnisse durch biochemische Untersuchungen, beispielsweise Sedimentationsessays. Dabei wird die Protein-Membran-Interaktion mit künstlichen Vesikeln und gereinigtem Protein in vitro, also außerhalb des Organismus, nachgewiesen. „Die Struktur von Mon1/Ccz1 hat eine einzigartige Architektur, die nach unserem aktuellen Wissensstand noch in keinem anderen Proteinkomplex nachgewiesen worden ist. Sie könnte als Blaupause für ein besseres Verständnis andere verwandter Regulatorproteine dienen. Wir wollen unsere erfolgreiche Zusammenarbeit fortsetzen“, sind sich Daniel Kümmel und Stefan Raunser einig.
Weitere Informationen und Finanzierung
Die Studie wurde im interdisziplinären Fachjournal „Proceedings of the National Academy of Sciences of the United States of America“ veröffentlicht. Neben Wissenschaftlern der WWU Münster und vom MPI Dortmund waren Forscher der Universität Osnabrück beteiligt. Die Deutsche Forschungsgemeinschaft unterstützte die Arbeiten im Rahmen des Sonderforschungsbereichs (SFB) 944 finanziell.
Originalpublikation
Björn U. Klink, Eric Herrmann, Claudia Antonia, Lars Langemeyer, Stephan Kiontke, Christos Gatsogiannis, Christian Ungermann, Stefan Raunser, Daniel Kümmel: Structure of the Mon1-Ccz1 complex reveals molecular basis of membrane binding for Rab7 activation. PNAS February 8, 2022 119 (6), DOI: 10.1073/pnas.2121494119
